OBJETIVO GENERAL
· Aprender la importancia y extracción de DNA de un hongo de importancia farmacéutica
OBJETIVOS ESPECÍFICOS
· Conocer los principios básicos para la extracción del material genético de un microorganismo
· Visualizar el material genético extraído con el uso de herramientas básicas de ingeniería genética
· Analizar el uso de éste material genético dentro de la industria farmacéutica
INTRODUCCIÓN
Los hongos no sólo intervienen en procesos de fermentación tales como producción de queso, cerveza o pan, sino que también tienen su aplicación en la Industria Farmacéutica ya que a partir de los hongos se fabrican, entre otros, antibióticos, drogas y tonificantes. El negocio de producción de antibióticos es uno de los más prominentes en la farmacia, pues alcanza ventas mundiales por más de 20,000 millones de dólares anuales (Lisdat et. al., 2008, pp. 655 ).
En ésta práctica obtuvimos el ADN genómico del hongo Agaricus bisporus para determinar su pureza y cuantificar su peso molecular comparado con un marcador genético del fago lambda. La importancia del hongo Agaricus bisporus no sólo se ve mostrada en el giro alimentario, dónde su consumo mundial es de alrededor de 0.44 kg per cápita incrementándose un 3.8% anualmente, sino también por los recientes estudios que se han realizado con el hongo intentando su transformación, véase p. ej. el trabajo de Mikosch et. al. 2001, donde se transformó el micelio de Agaricus bisporus por Agrobacterium tumefaciens, demostrándose la estabilidad de la transformación y siendo el primer trabajo dónde se transformaba A. bisporus.
La electroforesis en gel de agarosa es de las más utilizadas para analizar y caracterizar ácidos nucléicos de distintas procedencias. Los geles se comportan como un tamiz molecular y permiten separar moléculas cargadas en función de su tamaño y forma. Así, moléculas de DNA de diferente tamaño van a emigrar de forma distinta en una electroforesis en gel de agarosa. Y si el dicha electroforesis se aplican marcadores de peso molecular (en éste caso del fago λ) se puede conocer el tamaño aproximando del ADN de estudio (Padilla et. al., 2005) . El genoma del Agaricus bisporus se estima en 31 Mpb, organizado en 13 cromosomas(NCBI, 2002).
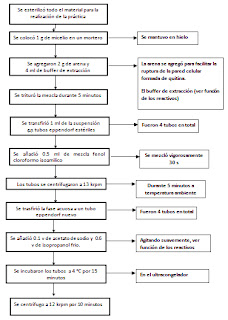
DESARROLLO EXPERIMENTAL
FUNCIÓN DE LOS REACTIVOS
Buffer de extracción: El cual tiene la función de disolver la bicapa lipídica así como proteger al ADN de la acción de enzimas nucleasas por acción del EDTA contenida en dicha solución que captura los iones magnesio y no permiten que actúen como cofactores de las nucleasas. Las altas concentraciones de NaCl se emplean para prevenir la contaminación de la muestra con polisacáridos que afectan la pureza del ADN, pudiendo inhibir la actividad de algunas enzimas como polimerasas, ligasas y endonucleasas de restricción; la base para la separación de los polisacáridos de los ácidos nucleicos, es su solubilidad diferencial en presencia de las altas concentraciones de NaCl los polisacáridos precipitan bajo la acción de fuerzas centrifugas.
ü Tris, tris (hidroximetil) aminometano (C4H11NO3; Mr = 121,14). Se utiliza como amortiguador porque es una sustancia inocua para la mayor parte de las proteínas. Su pka es 8,3 a 20 °C, lo que lo convierte en un amortiguador idóneo para el intervalo de pH entre 7 y 9.
ü Dodecilsulfato sódico(SDS) (C12H25NaO4S; Mr = 288,38). Es el agente disociador más habitual para desnaturalizar proteínas nativas en sus polipéptidos individuales. Cuando se calienta brevemente una mezcla de proteínas a 100 °C en presencia de SDS, el detergente que cubre el polipéptido alrededor de su eje central, manteniéndolo desplegado. En este proceso, las cargas intrínsecas del polipéptido son despreciables en comparación por las aportadas por el SDS. De este modo los polipéptidos se transforman después del tratamiento en estructuras con forma de bastón que poseen una densidad de carga uniforme en toda su longitud. La movilidad de estas proteínas es una función aproximadamente lineal del logaritmo de su masa molecular.
ü NaCl. Con el NaCl conseguimos producir el estallido de los núcleos para que queden libres las fibras de cromatina.
Fenol cloroformo isoamilico: la mezcla nos sirve para aislar proteínas
ü Isopropanol: Disminuye la solubilidad del DNA para que pueda precipitar de la solución. Puesto que las moléculas de la DNA son iónicas, debido a los grupos fosfatos, son altamente solubles en agua pero no soluble en solventes orgánicos. El isopropanol es de uso general precipitar ácidos nucleicos de soluciones acuosas.
ü Cloroformo. El cloroformo (o más usualmente fenol) es un desnaturalizador activo de proteínas que suprime la solubilidad de las proteínas en la preparación y las precipita. Puesto que el fenol y la solución salina amortiguadora no se mezclan, solo se requiere centrifugar la suspensión para separar las bases, permaneciendo el DNA (y el RNA) en la solución dentro de la fase acuosa de arriba y la proteína presente como un precipitado concentrado en la interfase.
Solución etanol agua: etanol se utiliza para precipitar la solución del ADN y para retirar sales
Buffer TE: protege al ADN de la degradación.
Agua mili Q .: agua ultra purificada, para proteger el ADN de la degradación
Buffer TAE: Este buffer sirve como amortiguador ya que es inocua para la mayor parte de las proteínas y también evita la acción de las nucleasas. El rojo Texas es un marcador fluorocromo unido de forma covalente a sondas de hibridación y a sondas de PCR
Buffer de carga: Este buffer le da peso a la muestra para que el ADN se precipite al fondo de los pozos asi como indicar el corrimiento de cada muestra debido al colorante
ü sacarosa: se usa en el buffer de carga para conferir peso a la muestra y esta no se disoerse en el buffer de corrimiento.
ü Azul de Bromofenol: El azul de bromofenol es un colorante que permite ver la corrida (corre como si fuera tuviera 500 pb de peso, es decir, está en el frente de la corrida), durante la electroforesis, controlando que la muestra no se caiga del gel o no se dirija a otro pocillo.
RESULTADOS
Electroforesis en gel de agarosa
Figura 1. Esquema de la cámara de electroforesis.
Figura 2. Electroforesis en gel de agarosa al 0.7% teñido con azul de bromofenol, se muestra el corrimiento del ADN extraído por diferentes equipos, al extremo derecho se encuentra el marcador de peso molecular (1kbp DNA extension ladder). Las bandas correspondientes al ADN del hongo para el equipo 6 se encuentran a 1,5 cm desde la base del pozo.
Figura 3. Bandeo del marcador de peso molecular 1kpb extension DNA ladder, corrido en un gel de agarosa al 1% con TAE 1X teñido con bromuro de etidio.
Tabla 1. Datos del bandeo para el marcador 1kpb extension DNA ladder en base a la figura 2.
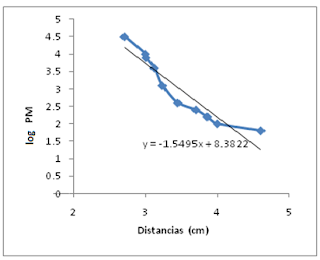
Figura 4. Relacion entre distancias del marcador dependientes del logaritmo del peso molecular.
Tabla 2. Comparación de los tamaños de genoma del Agaricus bisporus*
|
Tamaño teórico (kpb)
|
Estimación experimental (kpb)
|
|
31 000
|
1 142
|
Estimación de la concentración y pureza del ADN
Tabla 3. Resultados de la relación de absorbancias y las concentraciones* de las diferentes muestras de ADN.
DISCUSIÓN
Obtención de ADN
Para comenzar la discusión debemos tener en cuenta la importancia de los reactivos y los procedimientos a utilizar detallados anteriormente.
El buffer de extracción, o buffer de lisis, está compuesto básicamente de Tris que con un pKa de 8,1, es un regulador eficaz entre pH 7 y 9. Debido a su rango neutral, esto permite trabajar en la extracción sin desnaturalizar nuestros compuestos de interés. Sin embargo el Tris es sensible al calor lo cual nos obligó a trabajar en frío.
La primera fase de la extracción se lleva a cabo mediante el buffer de Tris y EDTA. El Tris como ya vimos protege de los cambios de pH y el EDTA tiene la facilidad de unirse a cationes divalentes como el calcio y el magnesio. Dado que estos iones ayudan a mantener la integridad de la membrana celular, la eliminación de ellos desestabiliza a la membrana; además de que su eliminación también ayuda a inactivar a las DNAsas que utilizan como cofactor al magnesio. En esta etapa el pH se mantiene alrededor de 8. No está confirmado aún pero se cree que el tris interactúa con los lipopolisacáridos en la membrana, desestabilizándola aún más. El efecto “detergente del SDS es bien conocido por lo que también es un componente fundamental del buffer de extracción ya que logra disolver los fragmentos de la membrana. Finalmente el cloruro de sodio aumenta la fuerza iónica del sistema y “rompe” los núcleos liberando la cromatina.
En el protocolo se detalla utilizar 4 mL del buffer de extracción pero finalmente utilizamos 6 mL. Realizando una analogía con una solución de cierta molaridad, el agregado de más buffer actúa como solvente ya que diluye la cantidad de ADN contenido por cada mL de solución. Este punto disminuyó la cantidad de muestra que colocamos en cada tubo Eppendorff.
La extracción, por razones de tiempo y espacio, no pudo realizarse en las condiciones expresadas en el protocolo, bajo flujo laminar y en un ambiente aséptico. Sin embargo tuvimos la precaución de “aislar” la mesa de trabajo lo máximo posible.
Figura 5. Etapas de una célula viva sometida a lisis. (A) Célula 5 minutos antes de la lisis. (B) 50 segundos después de la perfusión del buffer. (C) 1 minuto después de la perfusión: lisis de partida. (D) 6 minutos después de la perfusión: la lisis se ha completado.
Una de las técnicas utilizadas para obtener el DNA “purificado” o sin impurezas fue la del Salting-out. La misma consiste en precipitar las proteínas por saturación de la solución con sales neutras. Se sabe que cuando una sal (en nuestro caso acetato de sodio) se añade a una disolución de proteínas, se produce un incremento de la solubilidad de las mismas (solubilidad por salado o salting-in), lo que se explica por la presencia de contraiones que rodean a los grupos cargados de las proteínas, incrementando su solubilidad.
A medida que la fuerza iónica se eleva, por aumento en la concentración en la sal, se alcanza un máximo de solubilidad seguido de una disminución hasta que las proteínas precipitan. Las interacciones proteína-proteína se hacen más importantes, y por eso se produce su precipitación.
Figura 6 .Esquema de solubilidad de las proteínas vs concentración de la sal en la técnica Salting-out.
La técnica del Saltig-out puede realizarse varias veces para eliminar la mayor cantidad de proteínas posibles. En nuestro caso sólo la realizamos una vez lo que posiblemente influyó en el resultado de la estimación de pureza y concentración por espectrofotometría. Para la misma utilizamos el rango de luz UV debido a que los anillos presentes en las bases nitrogenadas del ADN tienen una absorbancia máxima a 260 nm. Por otra parte, la absorbancia máxima de las proteínas residuales se observa a los 280 nm.
Electroforesis en gel de agarosa
La electroforesis en gel de agarosa es de las más utilizadas para analizar y caracterizar ácidos nucleícos de distintas procedencias (Padilla, et al,2005). Para la realización de esta técnica, como se indico en el desarrollo experimental, se debe agregar un buffer de corrimiento que permite la circulación de la corriente en toda la cámara debido a los iones que contiene. La muestra se coloca en los pocillos junto con el buffer de carga con el objetivo de conferirle una mayor densidad y de esta manera precipite en el fondo del pozo y no se disperse flotando en el buffer de corrimiento; además se agregan colorantes para visualizar la muestra mientras corre por el gel, es este caso se uso azul de bromofenol.
Figura 7. Representación de la electroforesis en gel de agarosa esperado para la extracción de ADN genómico de hongo, el carril derecho representa el caso de que el DNA se haya sometido a DNAsas.
La razón por la cual esperar un corto desplazamiento del material genético es por su gran tamaño, pues datos del NCBI han reportado que el ADN genómico del Agaricus bisorus es de aproximadamente 31 Mpb.
Es importante el uso de marcadores de peso molecular (fragmentos de DNA de tamaño conocido) pues nos sirven para calcular el tamaño aproximado del DNA en estudio (Padilla, et al,2005). En los resultados de la electroforesis (ver figura 2) se distingue una banda a poca distancia del pozo, por lo cual se deduce que se trata del material genético extraído, incluso esta banda aparece de manera constante en el resto de los equipos. Otro aspecto a resaltar es que la banda antes mencionada se distingue a una distancia más corta (tomando como referencia la base del pozo) que la primer banda del marcador de peso molecular, por lo que se estima que el tamaño del ADN extraído es mayor a 40 kpb que corresponde a la primer banda del marcador (ver figura 3)
El uso de los marcadores no sólo es para comparar el corrimiento en el gel, pues debido a que se conoce el tamaño de los fragmentos que aparecen en el gel es posible establecer una correlación entre el desplazamiento del material genético y el tamaño del fragmento en pares de bases y de esa manera conocer el tamaño del ADN extraído, datos que se presentan en la tabla 2. El tamaño del AND extraído es de 1 142 kpb que representa casi la 27a parte del tamaño esperado (31 000 kpb), este resultado refleja es probable que estemos trabajando con un marcador de peso molecular que no utilizamos en la práctica por lo que no representa el verdadero peso del ADN extraído, además de que el bandeo del marcador utilizado fue corrido en un gel de agarosa al 1% y no al 0,7 % lo que evidencia más que los datos no sean precisos.
Espectrofotometría (concentración y pureza del ADN)
La estimación de pureza se realizó en base a la relación ADN/proteínas; según la bibliografía consultada, una buena extracción ronda en base a los 1.8-2.0. Analizando la figura 2 y la tabla 3 observamos que los resultados de nuestro equipo (N° 6) son coherentes en cuanto a distribución de las bandas y pureza estipulada (en el límite inferior de lo recomendado). Sin embargo el equipo 7 posee un bandeo en el gel mucho mejor, muy similar al marcador de referencia pero los datos de la tabla indican presencia de proteínas. Según la bibliografía consultada: Manual práctico de Bioquímica de la universidad de Quindio, Colombia la relación ADN/Proteínas del equipo 7 debía estar cercana a los 2.0. Una de las posibles causas de este error puede provenir de la dilución utilizada o el factor de dilución utilizado para realizar los cálculos(ver memoria de calculos)
El bandeo del ADN se ve tenue en la electroforesis, figura 2, esto se debe a que el ADN fue degradado, lo que se evidencia mas por el barrido que se observa a lo largo del gel hasta terminar con una mancha el final del corrimiento. Este resultado esta involucrado con la pureza y concentración de ADN.
Por lo que no se siguió adecuadamente el procedimiento para la extracción y/o que no se tuvieron los cuidados necesarios para mantener la integridad de ADN genómico, entre los errores detectados son:
· No haber trasferido de manera homogénea la suspensión de hongo sometido al buffer de extracción.
· En el momento de decantar haber perdido material genético por descuido.
· No ser cuidadosos con el material genético durante el secado y dejarlo momentáneamente expuesto al aire donde circulas muchas DNAsas.
· Uso inadecuado de los reactivos, por ejemplo el buffer de extracción contiene SDS que si bien produce la precipitación de gran parte de las proteínas, autores dicen que también precipita el ADN cromosómico, probablemente porque se producen asociaciones al azar entre las diferentes regiones de este ADN y así se forman agregados insolubles(Padilla, et al,2005), por lo que si se agrego este buffer de manera inadecuado pudo contribuir a tener una menor cantidad de ADN.
CONCLUSIONES
· Se aprendieron los principios básicos para llevar a cabo una extracción de ADN genómico en hongos.
· Se aprendió el fundamento teórico que justifica el uso de cada reactivo durante el protocolo de extracción de ADN genómico.
· Se verificó que el uso inadecuado de los reactivos en el procesos de obtención de ADN genómico afecta las concentración y pureza resultante.
· Se verificó que las condiciones bajo las que se realiza la extracción de ADN genómico influyen en la concentración y pureza del ADN, y que se evidencian con su visualización en el gel de agarosa.
· El procedimiento seguido para la obtención de ADN genómico de hongos es reproducible.
· Se comprobó que cambiar las condiciones de corrimiento (tiempo y voltaje) no influye demasiado en la visualización del ADN extraído.
· El ADN genómico no se encontraba intacto, lo cual se vio con la aparición de barrido en el gel de agarosa.
· El uso de marcadores de peso molecular es indispensable para estimar el tamaño del material genético obtenido.
· El uso de un colorante para el corrimiento de la electroforesis es útil para ubicar a nuestra muestra y así evitar que se salga del carro.
· Los métodos espectrofotométricos para obtener datos del factor de pureza y la electroforesis en gel de agarosa son métodos que nos permiten identificar el ADN extraído con la metodología seguida.
· Se comprendió la importancia de los hongos en la industria farmacéutica y la utilidad de la ingeniería genética para mejorar procesos de producción.
REFERENCIAS
-Alberts, B., Bray, B., Lewis, J., Raff, M., Roberts, K., y Watson, J. (2002). Biología molecular de la célula (3a ed.). Barcelona, España: Ediciones Omega. ISBN: 84-282-1011-X
-Blakebrough, N., Fiechter, A. and Ghose, T. K. (1979). Advances in Biochemical Engineering. Vol. II (1a Ed.). Berlin, Germany: Edit. Springer-Verlag. ISBN: 0-387-08990-X
-Crommelin y Sindelar (2002). Pharmaceutical biotechnology. 2da edición. Editorial Taylor & Francis Group.USA. p.p 44-46
-Kayser, O. and Müller, R. H. (2004). Pharmaceutical Biotechnology, Drug Discovery and Clinical Applications (1a Ed.). Weinheim, Germany: Edit. Wiley-VCH. ISBN: 3-527-30554-8
-Klefenz, H. (2002). Industrial pharmaceutical biotechnology (1a Ed.). Bornheim, Germany: Edit. Wiley-VCH. ISBNs: 3-527-60012-4
-Lisdat, F. and Renneberg, R. (2008). Advances in Biochemical Engineering/Biotechnology. Berlin, Germany: Edit. Springer-Verlag. ISBN: 978-3-540-75201-1
-Mikosch, TS., Lavrijssen, B., Sonnerberg, AS. y van Griensven, LJ. Transformation of the cultivated mushroom Agaricus bisporus (Lange) using T-DNA from Agrobacterium tumefaciens. Department of Genetics and Breeding, Mushroom Experimental Station, Horst, The Netherlands.Curr Gene.t 2001 Feb; 39(1):35-9.
-National Center for Biotechnology Information. http://www.ncbi.nlm.nih.gov . fecha de consulta: 03-sep-2011
-Novo, F. J. (2007). Genética Humana. Conceptos, mecanismos y aplicaciones de la Genética en el campo de la Biomedicina (1ª ed.). Madrid, España: Person Prentice-Hall. ISBN: 978-84-8322-359-8
-Padilla, C., Diez, J., Martínez, G., Bárcena, J. y García, C. (2005). Electroforesis de ácidos nucleicos en geles de agarosa. Aislamiento y caracterización electroforética de ADN plasmídico. Departamento de Bioquímica y Biología Molecular, Campus Universitario de Rabanales, Córdoba, España.
-Walsh, G. (2007). Pharmaceutical Biotechnology. Concepts and Applications (1a Ed.). England: John Wiley & Sons. ISBN: 978-0-470-01244-4
“Lo oí y lo olvidé, lo ví y lo entendí, lo hice y lo aprendí”
Confucio
Si eres estudiante de una universidad latinoamericana (incluido México), tal vez te pueda interesar ésta oferta laboral para dar asesorías online de ciencias a estudiantes:
http://farmupibi.blogspot.mx/2015/03/oferta-laboral.html
También puedes buscar la entrada en mi blog como "Oferta laboral".











No hay comentarios:
Publicar un comentario
Deja tu comentario, puede ser anónimo. Todos son bienvenidos.